Leki z komputera
Filip Gołębiowski | 2005-03-07
 Naukowcy wykorzystali metody komputerowe w celu identyfikacji cząsteczek, które mogły by służyć jako przyszłe leki na ciężkie schorzenie układu nerwowego. Zanalizowali in silico ponad 1,5 miliona małych cząsteczek w celu identyfikacji tych o najwyższym powinowactwie do białka biorącego udział w patogenezie choroby i mogących być potencjalnymi kandydatami na terapeutyk.
Naukowcy wykorzystali metody komputerowe w celu identyfikacji cząsteczek, które mogły by służyć jako przyszłe leki na ciężkie schorzenie układu nerwowego. Zanalizowali in silico ponad 1,5 miliona małych cząsteczek w celu identyfikacji tych o najwyższym powinowactwie do białka biorącego udział w patogenezie choroby i mogących być potencjalnymi kandydatami na terapeutyk.
Rodzinna postać stwardnienia zanikowobocznego (ang. familial amyotrophic lateral sclerosis, FALS) to dziedziczna, uwarunkowana genetycznie, forma choroby. Jest to poważne schorzenie centralnego i obwodowego układu nerwowego, powodujące degenerację nerwów motoryczno-ruchowych. Ta część układu nerwowego steruje ruchami i unerwia mięśnie. Objawami są osłabienie siły mięśni, drżenia i zanik mięśni. Efektem tego mogą być zaburzenia połykania, osłabienia kończyn, mięśni aparatu mowy. Najczęściej choroba ma przebieg łagodny i rozwija się latami.
Badacze, w swojej pracy, opierali się na wcześniejszych doniesieniach, mówiących iż FALS jest wywołana przez mutację w genie kodującym dysmutazę ponadtlenkową 1 (ang. superoxide dismutase 1, SOD-1). Jest to metaloenzym, odpowiedzialny w organiźmie za neutralizację szkodliwych anionorodników ponadtlenkowych (O2˙). Prowadzi ona przekształcenie dwóch takich rodników w nadtlenek wodoru i tlen cząteczkowy.
O2˙– + O2˙– + 2H+ → H2O2 + O2
reakcja katalizowana przez dysmutazę ponadtlenkową
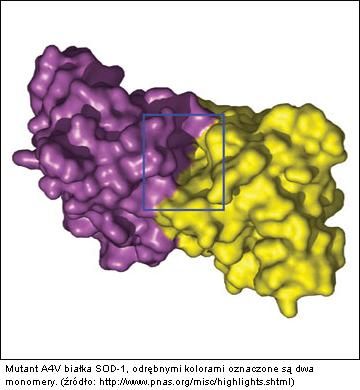 W miejscu aktywnym tego cytoplazmatycznego enzymu znajduje się jon miedzi i cynku. Jego chorobotwórcza, zmutowana forma zamiast dimeru tworzy nierozpuszczalne, toksyczne agregaty. Wpierw jednak musi zajść dysocjacja dimeru. Badacze doszli do wniosku, że jeśli udało by im się znaleźć związek, który byłby wstanie stabilizować zmutowany dimer, poprzez zapobieganie jego dysocjacji, to mogli by zapobiec tworzeniu się chorobotwórczych agregatów.
W miejscu aktywnym tego cytoplazmatycznego enzymu znajduje się jon miedzi i cynku. Jego chorobotwórcza, zmutowana forma zamiast dimeru tworzy nierozpuszczalne, toksyczne agregaty. Wpierw jednak musi zajść dysocjacja dimeru. Badacze doszli do wniosku, że jeśli udało by im się znaleźć związek, który byłby wstanie stabilizować zmutowany dimer, poprzez zapobieganie jego dysocjacji, to mogli by zapobiec tworzeniu się chorobotwórczych agregatów.
Autorzy pracy, Soumya Ray i jego współpracownicy, wykorzystali metody komputerowe w celu analizy ponad 1,5 miliona małocząsteczkowych związków chemicznych i wybrania najsilniej wiążących się do specyficznego miejsca dimeru, w taki sposób, by hamować dysocjację monomerów. Ta procedura pozwoliła naukowcom na identyfikację 100 cząsteczek które wiązały się ,według nich, najsilniej. Przeprowadzili oni potem badania in vitro, w celu sprawdzenia wyników analizy komputerowej. Okazało się, że spośród 100 cząsteczek 15, w sposób według nich znaczący, opóźniało agregację trzech wadliwych białek SOD-1 z mutacjami w miejscach Ala4Val, Gly93Ala, lub Gly85Arg. Znalezione przez nich małocząsteczkowe związki stabilizują wadliwe formy enzymu.
Ich praca ukazała się w magazynie Proceedings of the National Academy of Science (PNAS). Pokazuje jak wykorzystanie współczesnych metod komputerowych, modelowania molekularnego, umożliwia wstępne sprawdzenie siły wiązania ogromnej ilości związków, w celu znalezienia potencjalnych terapeutyków.
Źródła:
"Inhibition of aggregation of familial amyotrophic lateral sclerosis-linked superoxide dismutase 1 mutants by drug-like structural stabilizers" by Soumya S. Ray, Richard J. Nowak, Robert H. Brown, Jr., and Peter T. Lansbury, Jr.; Proc. Natl. Acad. Sci. USA Published online before print February 28, 2005
PNAS Article Highlights
Zdjęcie:
mutant A4V białka SOD-1, odrębnymi kolorami oznaczone są dwa monomery.
Pochodzi ze strony: PNAS
Komentarze
Kinga | 2006-07-20 00:00:00
Bardzo ciekawe tylko chyba nie dlamnie bo ja mam antrhopy muscular:(
