Pierwsze komercyjne badania diagnostyczne w Polsce przy wykorzystaniu mikromacierzy DNA
CBDNA | 2009-05-17
 Centrum Badań DNA z Poznania jest pierwszą instytucją w Polsce oferującą komercyjne badania diagnostyczne chorób i predyspozycji genetycznych przy wykorzystaniu mikromacierzy DNA. W testach diagnostycznych stosowane są mikromacierze typu SNP w technologii APEX, umożliwiające badanie setek fragmentów DNA i występujących w nich mutacji w jednym eksperymencie.
Centrum Badań DNA z Poznania jest pierwszą instytucją w Polsce oferującą komercyjne badania diagnostyczne chorób i predyspozycji genetycznych przy wykorzystaniu mikromacierzy DNA. W testach diagnostycznych stosowane są mikromacierze typu SNP w technologii APEX, umożliwiające badanie setek fragmentów DNA i występujących w nich mutacji w jednym eksperymencie.
"Wcześniej korzystały z niej jedynie ośrodki naukowe. My chcemy wprowadzić ją do rutynowej praktyki laboratoryjnej. Dlatego też nawiązaliśmy współpracę z estońską firmą ASPER Biotech, która opracowała i opatentowała technologię APEX microarray, umożliwiającą badanie setek fragmentów DNA i występujących w nich mutacji w jednym eksperymencie." - mówi Jacek Wojciechowicz, współzałożyciel Centrum. "Coraz więcej wiemy na temat wpływu różnych genów i ich dysfunkcji na etiologię różnych chorób, w tym przede wszystkim wrodzonych. Okazuje się ze podłoże wielu z nich jest bardzo złożone. Często dana choroba, np. rak piersi, nie jest powodowana mutacją w pojedynczym genie (to byłoby bardzo proste do zbadania), ale mogą odpowiadać za nią zmiany w wielu genach. Analiza wszystkich, za pomocą tradycyjnego sekwencjonowania czy PCR byłaby poważnym projektem badawczym wymagającym znacznych nakładów finansowych."
Zastosowanie mikromacierzy DNA typu SNP umożliwia analizę w pojedynczym eksperymencie nawet kilkuset mutacji i polimorfizmów odpowiedzialnych za rozwój chorób genetycznych. Mikromacierze DNA wykorzystywane są do poznawania mechanizmów chorobotwórczych oraz oceny ryzyka rozwoju chorób mających podłoże genetyczne. Technika ta umożliwia postawienie bardzo wczesnej i precyzyjnej diagnozy oraz dostosowanie leczenia do indywidualnego przypadku pacjenta.
Mikromacierze DNA typu SNP to niewielkie płytki, na które naniesiono wiele tysięcy sond, z których każda jest specyficzna tylko dla jednej mutacji lub polimorfizmu genetycznego. Płytka z badanym materiałem genetycznym trafia do komory hybrydyzacyjnej i zostaje poddana analizie w czytniku laserowym: na ekranie komputera badacz widzi setki tysięcy maleńkich kolorowych kropek, z których każda odpowiada jednej sondzie. Każda sonda identyfikuje określony lokus (SNP). Obserwując barwne punkty, badacze ustalają, jaki nukleotyd znajduje się w interesującym miejscu w sekwencji DNA, a tym samym czy zmiana ta ma charakter mutacji.
Mikromacierze typu SNP w technologii APEX
Technologia APEX (Arrayed Primer Extension) opiera się na wykorzystaniu ddNTP (dideoksy nukleotydów) znakowanych fluorochromami. Jest to modyfikacja metody single nucleotide primer extension, z tym że w tym przypadku sondy unieruchomione są na macierzy. Każdy rodzaj ddNTP znakowany jest innym barwnikiem.
W pierwszym etapie wykonuje się PCR powielając interesujące nas sekwencje genów. W reakcji wykorzystuje się domieszkę dUTP, tak by po zakończonej reakcji za pomocą enzymu UNG (Uracylo N-glikozylaza) doprowadzić do częściowej fragmentacji materiału.
W drugim etapie następuje hybrydyzacja pofragmentowanego DNA pacjenta z mikromacierzą. Na powierzchni płytki w ściśle określonych punktach umieszczone są oligonukleotydowe sondy. Po hybrydyzacji sondy z komplementarną sekwencja badanego DNA następuje 3 etap w którym wykorzystuje się ddNTPs.
Polimeraza DNA użyta w reakcji dobudowuje pojedynczy ddNTP do końca każdej sondy znajdującej się na macierzy. Sondy są tak zaprojektowane, aby dobudowywany nukleotyd był tym interesującym nas SNP świadczącym o obecności lub braku mutacji. Dobudowanie odbywa się na zasadzie komplementarności z nukleotydem w sekwencji DNA pacjenta, a więc bardzo specyficznie. W zależności, jaki ddNTP zostaje dołączony do końca sondy emituje on inną długość światła pod wpływem światła lasera.
Ostatnim etapem jest detekcja polegająca na wzbudzeniu płytki kolejno 4 różnymi długościami światła laserowego i odczytanie sygnałów pochodzących z poszczególnych punktów macierzy. W wyniku pełnego odczytu płytki a następnie analizę i przetworzenie programowe jesteśmy w stanie identyfikować poszczególne, pojedyncze nukleotydy znajdujące się w interesujących nas miejscach genomu.
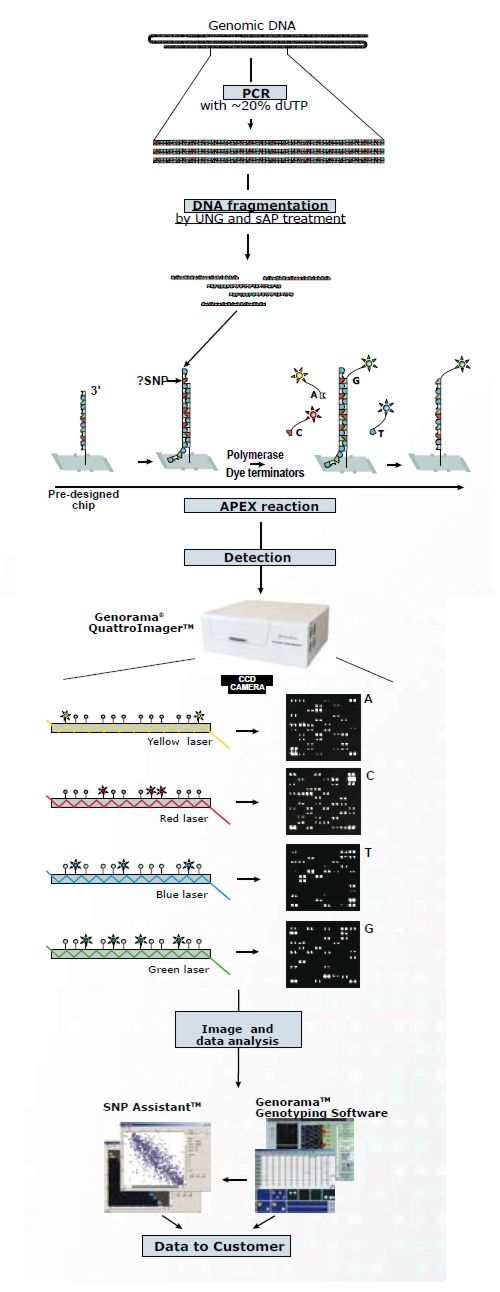
Schemat mikromacierzy w technologii APEX
Innowacyjność technologii APEX
Dotychczas technologia mikromacierzy wykorzystywana była w Polsce głównie do celów naukowych, a najczęściej stosowane - mikromacierze ekspresyjne wykorzystywane były w badaniach różnicy w ekspresji genów np.: w tkance pacjenta zdrowego oraz w tkance nowotworowej, czy badaniach nad RNAi (wyciszaniem genów).
Mikromacierze DNA w technologii APEX - to zupełnie nowa technologia umożliwiająca identyfikacje mutacji punktowych i polimorfizmy w sekwencji DNA. Technologia opracowana przez Asper Biotech Ltd., pozwala na analizę nawet kilkuset mutacji punktowych w jednym eksperymencie. Prowadzi to do znaczącego obniżenia kosztów analizy w przeliczeniu na jedną mutację (koszt analizy jednej mutacji wynosi ok 5zł). Ponadto dzięki mikromacierzom DNA możliwe stało się badanie wszystkich znanych mutacji odpowiedzialnych za daną jednostkę chorobową, a nie badanie tylko kilku najczęstszych mutacji jak to miało miejsce dotychczas.
"Chcemy również dostosować oferowane testy do wymagań polskiej populacji. Jeśli uznamy, że określonej mutacji brakuje, wprowadzimy ją do mikromacierzy. Planujemy także opracowywać nowe mikromacierze wspólnie z Estończykami. W tej chwili pracujemy nad mikromacierzą do diagnostyki predyspozycji do raka jelita grubego." - mówi Jacek Wojciechowicz. "Dzięki tej innowacyjnej technologii możliwe stało się wykonywanie kompleksowych badań w kierunku predyspozycji do chorób warunkowanych genetycznie, dzięki którym pacjent otrzymuje bardziej pewny i informatywny wynik badania."
Centrum jako pierwsze Laboratorium w kraju wprowadziło innowacyjną technologię DPO-PCR (ang. dual priming oligonuclotide PCR) umożliwiającą identyfikację kilkunastu różnych patogenów w jednym badaniu. Ta nowatorska technologia przyczyniła się do zaoferowania paneli badań diagnostycznych – test na sepsę (identyfikacja 65 patogenów wraz z określeniem lekooporności, panel Uro-Genital 21 – identyfikacja 21 patogenów dróg moczowo-płciowych. Urogenital to panele testów, które powstały z myślą o kobietach będących w ciąży lub je planujące. Wykrywają one najczęstsze zakażenia układu moczowo-płciowego, stanowiące największe zagrożenie dla kobiety w ciąży oraz jej dziecka przed, w trakcie i po porodzie, panel Pneumototal – identyfikacja 13 wirusów i 6 bakterii powodujących zakażenia górnych dróg oddechowych, i inne). Głównym celem jest oferowanie kompleksowych testów diagnostycznych.
Centrum Badań DNA
Centrum Badań DNA obecnie oferuje 16 testów diagnostycznych w technologii mikromacierzy DNA, a wśród nich:
Rak piersi i jajnika - szacuje się, że za około 5-10% przypadków raka piersi i jajnika odpowiedzialne są mutacje odziedziczone. Kluczowymi genami związanymi z dziedziczną odmiana tych nowotworów są BRCA1 i BRCA2. Przyczyną wysokiej umieralności kobiet w Polsce z powodu raka piersi i jajnika jest słaba profilaktyka, w tym zbyt mała liczba badań genetycznych. Dzięki testom wykonywanym w Centrum Badań DNA identyfikuje się 85 mutacji w 6 genach. To największy tego typu panel badań Polsce.
Mukowiscydoza - jest to najczęściej występująca choroba genetyczna (1 zachorowanie na 2.500 urodzeń). Przyczyną choroby są mutacje w genie CFTR. Dzięki genetyce molekularnej chorobę można rozpoznać już we wczesnym okresie ciąży. W Polsce średni wiek dziecka w chwili postawienia diagnozy to około 4-5 lat (w Europie Zachodniej są to pierwsze miesiące życia). Test wykonywany w Centrum Badań DNA wykrywa 254 najistotniejsze mutacje genu CFTR odpowiedzialne za występowanie mukowiscydozy.
Talasemia - jest to ilościowe zaburzenie syntezy hemoglobiny, spowodowane wrodzonym defektem biosyntezy łańcuchów globiny. Test umożliwia identyfikacje 90 mutacji w 3 genach kodujących łańcuchy beta i delta globiny.
Choroby Żydów Aszkenazyjskich - występowanie chorób w określonych grupach etnicznych spowodowane jest kulturową regułą endogamii. Centrum Badań DNA jest jedyna firmą w Polsce, która w tak szerokim panelu diagnozuje najczęstsze mutacje genów powiązane miedzy innymi z takimi schorzeniami jak: mukowiscydoza, zespół Blooma, choroba Niemanna-Picka czy choroba Taya-Sachsa. Test umożliwia wykrycie 77 mutacji w 22 genach.
Retinopatia (barwnikowa dziedziczona autosomalnie recesywnie i autosomalnie dominująco) - retinopatia barwnikowa (zwyrodnienie barwnikowe siatkówki) to choroba oczu, która dotyka około miliona osób na całym świecie. Występują dwa typy dziedziczenia. Typ w znaczącej części decyduje o przebiegu choroby, dlatego bardzo istotne dla pacjenta jest jego ustalenie. Panele umożliwiają oznaczenie 491 mutacji w 17 genach dla RP dziedziczonej autosomalnie recesywnie oraz 353 mutacji w 13 genach dla RP dziedziczonej autosomalnie dominująco.
Źródło: materiały CBDNA
